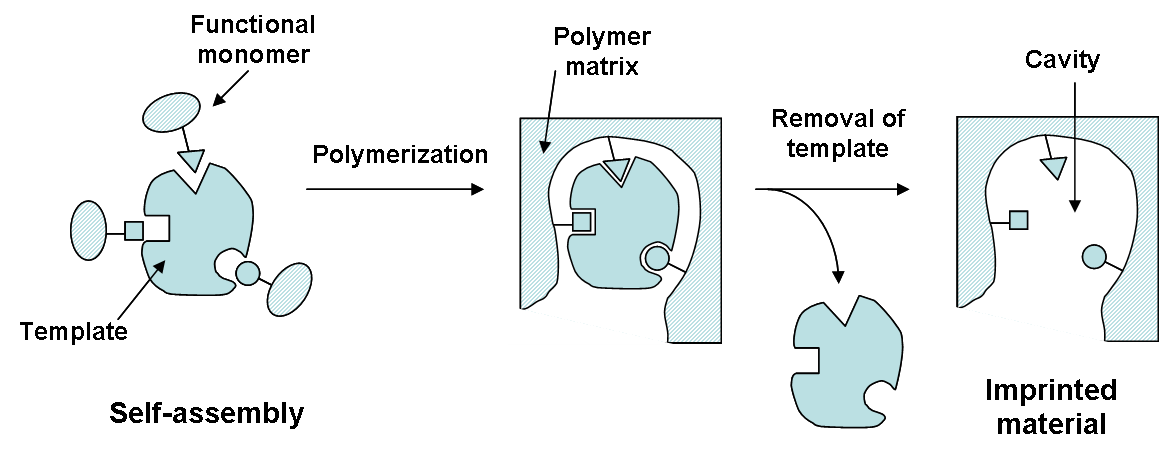
分子印记聚合物
分子印迹聚合物(MIP) 是一种是一种使用分子印迹技术加工而成的聚合物,该技术在聚合物基质中留下空腔,对选定的“模板”分子具有亲和力。制作 MIP 的过程通常涉及在模板分子存在下引发单体聚合,然后将模板分子提取出来,留下与之互补的空腔。这些聚合物对原始分子有亲和力,并且已经用于化学分离、催化或分子传感器等应用领域。相关研究可以追溯到20世纪30年代。
分子印迹技术的最新技术和观点
分子印记是在固体或凝胶内产生印记的过程,其大小、形状和电荷分布对应于(通常在聚合过程中存在的)模板分子。合成的受体能够以高亲和力和特异性结合目标分子。聚合物与模板之间的相互作用类似于抗体与抗原之间的相互作用,包括静电相互作用、氢键、范德华力和疏水相互作用。
与天然存在的受体相比,人工受体的最大优势之一是分子设计的自由度。它们的框架不限于蛋白质,可以使用各种骨架(例如,碳链和稠合芳环)。因此,稳定性、灵活性和其他特性可以根据需要自由调节。甚至在自然界中未发现的官能团也可用于这些合成化合物中。此外,必要时,可以通过使用适当的官能团来提供对外部刺激(光辐射、pH变化、电场或磁场等)的响应活性。
在分子印迹过程中,需要
- 模板
- 功能单体
- 交联剂
- 自由基或其他聚合引发剂
- 致孔溶剂
- 萃取溶剂
根据聚合方法和最终聚合物形式,可能需要避免使用某些方法或者试剂。1
主要有两种方法可以制造这些专用聚合物。第一种是自组装,即将MIP的所有元素结合起来形成聚合物,可能还会利用分子间作用形成带有结合模板分子的交联聚合物。第二种方法是先将模板分子共价连接到单体上,聚合后,再将模板分子裂解下来。 2选择性受印迹聚合物合成中使用的交联剂的种类和用量的影响很大。选择性还取决于目标分子和单体官能团之间的共价和非共价相互作用。功能单体的选择也对模板和底物互补相互作用有很大影响。 3在印迹聚合物中,交联剂具有三个主要功能:首先,交联剂可以控制聚合物基质的形态。其次,它可以稳定印迹结合位点。最后,它可以赋予聚合物基体机械稳定性。从聚合的角度来看,通常优选高交联比的聚合物,以便获得永久多孔材料,同时具有足够的机械稳定性。
自组装方法的优势在于它形成了更自然的结合位点,并且可以灵活选择单体。共价方法通常可以高产率地得到同质结合位点,但需要首先合成衍生化的印记分子,并且可能无法模仿可能存在于其他地方的“自然”条件。 4近年来,学术界和工业界对分子印迹技术的兴趣迅速增加。聚合方法方面取得了重大进展,这些方法可以产生具有相当好的结合特性的 MIP 形式,以期提高性能或适应所需的最终应用,例如珠状、薄膜或纳米粒子。
目前,在实际应用中限制 MIP 性能的主要问题是缺乏简单而稳健的方法地合成满足应用所需的 MIP。MIP 的第一种聚合方法是“本体”或溶液聚合。这是从事相关工作的课题组最常用的技术,因为它简单且功能多样。该方法使用具有低介电常数的有机溶剂,混合所有组分(模板、单体、溶剂和引发剂),然后聚合;将所得聚合物块粉碎、脱离模板、压碎和筛分以获得形状不规则且尺寸在 20 - 50 µm之间的颗粒。根据目标(模板)类型和 MIP 的最终应用,MIP 可以制成不同的形式,例如纳米/微米球形颗粒、纳米线和薄膜或膜。聚合物可以用不同的方法制备,如本体聚合、沉淀聚合、乳液聚合、悬浮聚合、分散聚合、凝胶聚合和多步溶胀聚合。 MIP 领域的大多数研究人员都在使用启发式技术(例如分层印记法)制作 MIP。该技术首次被 Sellergren 等人用于制作 MIP 5印迹小目标分子。Nematollahzadeh 等人 6开发了一种名为聚合填充床的通用技术,使用二氧化硅多孔颗粒进行蛋白质识别和捕获,获得结构分层的高容量蛋白质印迹多孔聚合物珠。
固相合成
固相分子印迹可以作为传统体印迹的替代方法,可用于产生水溶性纳米粒子。 7 8顾名思义,该技术需要在聚合之前将目标分子固定在固体支持物上。这类似于肽的固相合成。固相兼作亲和力分离基质,可以去除低亲和力的 MIP 并克服许多之前的 MIP 的限制:
- 从固定的模板分子中分离 MIPs 的过程大大简化。
- 结合位点更均匀,模板分子不会被困在聚合物基质中。
- MIP 可以在合成后官能化(同时附着在固相上),而不会显着影响结合位点。
- 固定化的模板可以重复使用,降低了 MIP 合成的成本。
通过这种方法合成的 MIP 纳米粒子已在各种诊断测定和传感器中得到应用。 9 10 11
分子建模
分子建模已成为 MIP 设计和分析中的一种便捷选择,结合一系列不同的技术,它可以用于快速选择单体和优化聚合物组。 12 13分子建模在这方面的应用通常归功于Sergey Pletsky和他的访问学生 Sreenath Subrahmanyam,他们开发了一种方法,可以使用分子力学方法针对给定的目标或模板自动筛选大型单体数据库14 15 。 16近年来,技术进步允许通过量子力学分子模型更有效地分析单体-模板相互作用,提供更精确的结合能计算。 17分子动力学也已应用于聚合前系统和生成的聚合物的更详细分析 18 19 20,与只考虑单体-模板相互作用相比,考虑了更多的系统组分(引发剂、交联剂、溶剂)的预测更成功。 21 22分子建模、特定分子动力学和不太常见的粗粒度技术, 23通常也可以集成到更大的理论模型中,结合热力学分析和动力学数据用于压印聚合物块体和 MIP 纳米粒子的介观分析。 24 25
应用
MIP 应用的利基领域是传感器和分离。尽管目前分子印迹的总体状况良好,但迄今为止仍然难以商业化。尽管如此,目前已有大量关于分子印迹的专利(根据Scifinder数据库截至 2018 年 10 月共有1035 项专利)。
分子印迹聚合物技术制备快速且成本较低,在化学、生物学和工程的许多领域都有应用,特别是用于传感器的亲和材料, 26检测化学品、抗菌剂和染料、食品中的残留物、固相萃取吸附剂、结合测定、人工抗体、色谱固定相、催化、药物开发和筛选以及化学反应中的副产物。 27分子印迹聚合物具有高度特异性的微腔结合位点,提供了广泛的提取能力。 28 29该技术在分析化学中有作为实用固相萃取方法应用的前景。 30 分子印迹聚合物技术能够更便宜、更容易地生产抗体/酶样结合位点,有望成为医学研究和应用中的一个有价值的突破。 31可能的医学应用包括“控释药物、药物监测装置和生物受体模拟物”。 32除此之外,MIP 在食品科学的发展和应用方面也有广阔的前景。 33 34
与特定抗体相比,MIP 的结合活性可能较低,尽管也已有报道称 MIP 的性能与商业生产的抗体相当或更好。 35 36这为 MIP 带来了从高效提取到制药/医疗用途的广泛的应用。 37 38与蛋白质结合位点相比,MIP 具有许多优势。蛋白质的纯化、变性(pH、热、蛋白水解)困难且昂贵,并且难以固定以供重复使用。合成聚合物便宜、易于合成,并可以结合复杂的合成侧链。特殊的侧链能够拥有更高的亲和力、选择性和特异性。
分子印迹分析可以说体现了分子印迹聚合物作为用于诊断应用的替代亲和试剂的最大潜力,因为它们具有与抗体相当(并且在某些方面更优越)的性能。因此,自 Vlatakis 等人的开创性工作以来,许多研究都集中在分子印迹分析 (MIA) 的开发上。 1993 年,首次引入了术语“分子印迹测定”。利用 MIP 代替抗体的配体结合测定的最初工作是使用放射性标记的 MIA,但是该领域现在已经发展到包括许多测定形式,例如荧光 MIA、酶联 MIA 和分子印迹纳米颗粒测定 (MINA)。 39
分子印迹聚合物也已用于从细胞裂解物中富集低丰度磷酸肽, 40优于目前的黄金标准——二氧化钛 (TiO2) 富集。
历史
在 1931 年发表的一篇论文中, 41 Polyakov 报道了在干燥新制备的二氧化硅过程中不同溶剂(苯、甲苯和二甲苯)的存在对二氧化硅孔结构的影响。当H2SO4被用作聚合引发剂(酸化剂)时,表面积(例如负载容量)与相应溶剂的分子量之间呈正相关。后来,在 1949 年,Dickey 报道了硅酸钠在四种不同染料(即甲基、乙基、正丙基和正丁基橙)存在下的聚合反应。随后去除染料,在再结合实验中发现,在任何这些“模板分子”存在的情况下制备的二氧化硅会优先于其他三种染料结合模板分子。这项工作发表后不久,几个研究小组开始使用 Dickey 方法制备特定的吸附剂。默克公司为尼古丁过滤器申请了专利, 42过滤器含有尼古丁印迹二氧化硅,能够比非印迹二氧化硅多吸附 10.7% 的尼古丁。该材料旨在用于香烟、雪茄和烟斗过滤器。这项工作出现后不久,分子印迹引起了科学界的广泛兴趣,1931-2009 年期间该领域发表了 4000 篇原始论文(来自 Scifinder)。然而,通常分子印迹技术只在靶向分子量 <1000 的小分子时才有效。 43因此,在下面的小节中,分子印迹聚合物被分为两类,分别为小模板和大模板。
生产限制
新型 MIP 的生产面临该领域独有的挑战。分子印记聚合物的基材各不相同,因此需要不同的单体和交联剂组合才能在特定基材上形成印迹聚合物。第一个也是较小的挑战是如何选择那些将产生与底物分子的官能团互补的结合单体。例如,选择完全疏水的单体来印上高度亲水的基材一般是不可取的。在创建新的 MIP 之前,需要考虑这些注意事项。分子建模可用于预测模板和单体之间的有利相互作用,从而实现智能单体选择。
更麻烦的是,一旦聚合物在周围形成,MIP 的产量受到从 MIP 上清洗模板的能力的限制。 44在创建 MIP 时,必须在完全移除原始模板和损坏基板结合腔之间做出折衷。结合腔损坏通常是由强力移除方法造成的,包括空腔塌陷、变形结合点、模板移除不完全和空腔破裂。
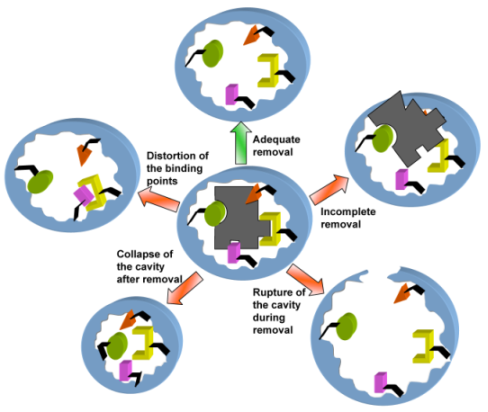 |
|---|
| 分子印迹聚合物模板去除过程面临的挑战 |
模板移除
在过去十年中,MIP 生产的大部分发展成果都是新聚合技术,这些聚合技术试图控制单体的排列,从而控制聚合物结构。然而,在模板聚合后从 MIP 中有效去除模板方面进展甚微。模板去除过程现在是 MIP 生产中成本效率最低且最耗时的过程。 45
目前有几种不同的提取方法用于模板去除。这些已分为 3 大类:溶剂萃取、物理辅助萃取以及亚临界或超临界溶剂萃取。
溶剂萃取
- 索氏提取自发明以来,一直是使用有机溶剂的标准提取方法。该技术将 MIP 颗粒放入提取室内的筒中,然后将提取溶剂倒入与提取室相连的烧瓶中。将溶剂加热并在柱内冷凝,从而接触 MIP 颗粒并萃取模板。 46该技术的主要优点是用新鲜的萃取溶剂反复洗涤 MIP 颗粒,由于使用热溶剂有利于溶解,完成后无需过滤即可收集 MIP 颗粒,设备价格适中,用途广泛,可以适用于几乎任何聚合物基体。 47主要缺点是提取时间长,使用的有机溶剂量大,对温度敏感的聚合物可能会分解,该技术的静态性质不利于溶剂流过 MIP,并且难以实现自动化。 47
- 孵育是将 MIP 浸入溶剂中,溶剂会导致聚合物溶胀,有利于模板从聚合物中解离。一般这种方法在温和的条件下进行,聚合物的稳定性不受影响。然而,与索氏提取技术类似,此方法也非常耗时。 48
- 固相模板将模板分子固定在玻璃珠等固体支持物上,可以轻松从模板中去除 MIP。在冷洗去除未反应的单体和低亲和力聚合物后,可以添加热溶剂以破坏结合并收集高亲和力 MIP。 49 50
物理辅助提取
- 超声波辅助提取 (UAE)通过空化的过程发挥左右,该过程在液体中形成小气泡,对固体颗粒造成机械侵蚀。这导致局部温度和压力升高,有利于溶剂和模板分子的溶解扩散、渗透传输。 51 52
- 微波辅助萃取 (MAE)使用微波直接与分子相互作用,造成离子传导和偶极子旋转。可以使提取迅速发生,但是,如果聚合物对热敏感,则必须小心避免温度过高。当该技术与强有机酸一起使用时,效果最好,但是,这也可能导致部分 MIP 降解。 53这种方法显着减少了提取模板所需的时间,降低了溶剂成本,是一种清洁的技术。 54
- 机械方法:一项研究表明,微接触分子印迹方法可以从模板中机械去除目标(大生物分子、蛋白质等)。该技术与生物传感器应用相结合,有望用于生物技术、环境和医学应用。 55
亚临界或超临界溶剂萃取
- 亚临界水 (PHWE) 方法在高温(100–374 °C) 高压(10–60 bar)下使用水作为溶剂。水在高温下极性显著降低,使得该条件下水可以溶解各种极性、离子性或非极性化合物。在高温高压条件下,水的粘度与表面变张力降低,也有利于扩散。此外,高温能破坏分子间作用力,例如模板与基质间的偶极-偶极相互作用、范德华力、氢键等,有利于解离的发生。
- 超临界 CO2 (SFE) 是一种绿色的提取方法,使用超临界 CO2 作为溶剂。超临界 CO2 的临界点为 31.1 °C 和 73.8 bar,此时 CO2 的密度与液体相近,但是具有气体的扩散性。超临界 CO2 的极性较低,因此对于极性化合物的溶解度较低,但是对于非极性化合物的溶解度较高。此外,超临界 CO2 的粘度低,有利于扩散。超临界 CO2 的温度较低,因此对于热敏感的聚合物有利。
参见
- 人工酶
外部链接
参考文献
本文采用CC-BY-SA-4.0协议发布,来自我创建的Wikipedia条目分子印记聚合物,由于个人水平限制,本条目难免会有知识谬误以及描述不清等情况,欢迎读者在维基条目中编辑修改或对本网站在GitHub上的源代码提供Pull request,也欢迎在评论区多多指教。
-
Sellergren B. Molecularly Imprinted Polymers: Man-made mimics of antibodies and their applications in analytical chemistry. Amsterdam: Elsevier. 2001. ↩
-
Tse Sum Bui B, Haupt K. Molecularly imprinted polymers: synthetic receptors in bioanalysis. Analytical and Bioanalytical Chemistry. November 2010, 398 (6): 2481–92. PMID 20845034. S2CID 20907385. doi:10.1007/s00216-010-4158-x. ↩
-
“Characteristic and Synthetic Approach of Molecularly Imprinted Polymer” Int. J. Mol. Sci. 2006, 7, 155–178 ↩
-
Mosbach H. Molecularly Imprinted Polymers and Their Use in Biomimetic Sensors. Chem Rev. 2000. ↩
-
Sellergren B, Buechel G. A porous, molecularly imprinted polymer and preparation. PCT Int. Appl. 1999. ↩
-
Nematollahzadeh A, Sun W, Aureliano CS, Lütkemeyer D, Stute J, Abdekhodaie MJ, et al. High-capacity hierarchically imprinted polymer beads for protein recognition and capture. Angewandte Chemie. January 2011, 50 (2): 495–8. PMID 21140388. doi:10.1002/anie.201004774. ↩
-
Canfarotta F, Poma A, Guerreiro A, Piletsky S. Solid-phase synthesis of molecularly imprinted nanoparticles. Nature Protocols. March 2016, 11 (3): 443–55. PMID 26866789. S2CID 20963528. doi:10.1038/nprot.2016.030. ↩
-
Poma A, Guerreiro A, Whitcombe MJ, Piletska EV, Turner AP, Piletsky SA. Solid-Phase Synthesis of Molecularly Imprinted Polymer Nanoparticles with a Reusable Template - “Plastic Antibodies”. Advanced Functional Materials. June 2013, 23 (22): 2821–2827. PMC 4746745 可免费查阅. PMID 26869870. doi:10.1002/adfm.201202397. ↩
-
Xu J, Prost E, Haupt K, Bui TS. Direct and Sensitive Determination of Trypsin in Human Urine Using a Water-Soluble Signaling Fluorescent Molecularly Imprinted Polymer Nanoprobe. Sensors and Actuators. 2017, 258: 10–17. doi:10.1016/j.snb.2017.11.077. ↩
-
Smolinska-Kempisty K, Guerreiro A, Canfarotta F, Cáceres C, Whitcombe MJ, Piletsky S. A comparison of the performance of molecularly imprinted polymer nanoparticles for small molecule targets and antibodies in the ELISA format. Scientific Reports. November 2016, 6: 37638. Bibcode:2016NatSR…637638S. PMC 5121619 可免费查阅. PMID 27883023. doi:10.1038/srep37638. ↩
-
Smolinska-Kempisty K, Ahmad OS, Guerreiro A, Karim K, Piletska E, Piletsky S. New potentiometric sensor based on molecularly imprinted nanoparticles for cocaine detection. Biosensors & Bioelectronics. October 2017, 96: 49–54. PMID 28472729. doi:10.1016/j.bios.2017.04.034. hdl:2381/39964 可免费查阅. ↩
-
Sullivan, Mark V.; Dennison, Sarah R.; Archontis, Georgios; Reddy, Subrayal M.; Hayes, Joseph M. Toward Rational Design of Selective Molecularly Imprinted Polymers (MIPs) for Proteins: Computational and Experimental Studies of Acrylamide Based Polymers for Myoglobin (PDF). The Journal of Physical Chemistry B. 5 July 2019, 123 (26): 5432–5443. PMID 31150581. S2CID 172137800. doi:10.1021/acs.jpcb.9b03091. ↩
-
Cowen T, Karim K, Piletsky S. Computational approaches in the design of synthetic receptors - A review. Analytica Chimica Acta. September 2016, 936: 62–74. PMID 27566340. doi:10.1016/j.aca.2016.07.027. ↩
-
Subrahmanyam, Sreenath; Piletsky, Sergey A; Piletska, Elena V; Chen, Beining; Karim, Kal; Turner, Anthony P.F. ‘Bite-and-Switch’ approach using computationally designed molecularly imprinted polymers for sensing of creatinine11Editors Selection. Biosensors and Bioelectronics. 2001–2012, 16 (9–12): 631–637. PMID 11679238. doi:10.1016/S0956-5663(01)00191-9 (英语). ↩
-
Piletsky SA, Karim K, Piletska EV, Day CJ, Freebairn KW, Legge C, Turner AP. Recognition of ephedrine enantiomers by molecularly imprinted polymers designed using a computational approach. Analyst. 2001, 126 (10): 1826–1830. Bibcode:2001Ana…126.1826P. S2CID 97971902. doi:10.1039/b102426b. ↩
-
Khan MS, Pal S, Krupadam RJ. Computational strategies for understanding the nature of interaction in dioxin imprinted nanoporous trappers. Journal of Molecular Recognition. July 2015, 28 (7): 427–37. PMID 25703338. S2CID 23551720. doi:10.1002/jmr.2459. ↩
-
Wagner, Sabine; Zapata, Carlos; Wan, Wei; Gawlitza, Kornelia; Weber, Marcus; Rurack, Knut. Role of Counterions in Molecularly Imprinted Polymers for Anionic Species. Langmuir. 2018-06-12, 34 (23): 6963–6975. ISSN 0743-7463. PMID 29792030. doi:10.1021/acs.langmuir.8b00500 (英语). ↩
-
Golker K, Nicholls IA. The effect of crosslinking density on molecularly imprinted polymer morphology and recognition. European Polymer Journal. 2016, 75: 423–430. doi:10.1016/j.eurpolymj.2016.01.008. ↩
-
Cowen T, Busato M, Karim K, Piletsky SA. In Silico Synthesis of Synthetic Receptors: A Polymerization Algorithm. Macromolecular Rapid Communications. December 2016, 37 (24): 2011–2016. PMID 27862601. doi:10.1002/marc.201600515. hdl:2381/40379 可免费查阅. ↩
-
Sobiech M, Żołek T, Luliński P, Maciejewska D. A computational exploration of imprinted polymer affinity based on voriconazole metabolites. The Analyst. April 2014, 139 (7): 1779–88. Bibcode:2014Ana…139.1779S. PMID 24516859. doi:10.1039/c3an01721d. ↩
-
Piletska EV, Guerreiro A, Mersiyanova M, Cowen T, Canfarotta F, Piletsky S, et al. Probing Peptide Sequences on Their Ability to Generate Affinity Sites in Molecularly Imprinted Polymers. Langmuir. January 2020, 36 (1): 279–283. PMID 31829602. S2CID 36207119. doi:10.1021/acs.langmuir.9b03410. ↩
-
Levi L, Raim V, Srebnik S. A brief review of coarse-grained and other computational studies of molecularly imprinted polymers. Journal of Molecular Recognition. 2011, 24 (6): 883–91. PMID 22038796. S2CID 30296633. doi:10.1002/jmr.1135. ↩
-
Srebnik S. Theoretical investigation of the imprinting efficiency of molecularly imprinted polymers. Chemistry of Materials. 2004, 16 (5): 883–888. doi:10.1021/cm034705m. ↩
-
Cowen T, Karim K, Piletsky SA. Solubility and size of polymer nanoparticles. Polymer Chemistry. 2018, 9 (36): 4566–4573. doi:10.1039/C8PY00829A. hdl:2381/43254 可免费查阅. ↩
-
Delaney TL, Zimin D, Rahm M, Weiss D, Wolfbeis OS, Mirsky VM. Capacitive detection in ultrathin chemosensors prepared by molecularly imprinted grafting photopolymerization. Analytical Chemistry. April 2007, 79 (8): 3220–5. PMID 17358046. doi:10.1021/ac062143v. ↩
-
Lok CM, Son R. Application of molecularly imprinted polymers in food sample analysis – a perspective (PDF). International Food Research Journal. 2009, 16: 127–140. ↩
-
Wulff G, Sarhan A. Über die Anwendung von enzymanalog gebauten Polymeren zur Racemattrennung.. Angewandte Chemie. April 1972, 84 (8): 364. Bibcode:1972AngCh..84..364W. doi:10.1002/ange.19720840838. ↩
-
Wulff G, Sarhan A, Zabrocki K. Enzyme-analogue built polymers and their use for the resolution of racemates.. Tetrahedron Letters. 1973, 14 (44): 4329–32. doi:10.1016/S0040-4039(01)87213-0. ↩
-
Olsen J, Martin P, Wilson ID. Molecular imprints as sorbents for solid phase extraction: potential and applications. Anal. Commun. 1998, 35 (10): 13H–14H. doi:10.1039/A806379F. ↩
-
Ertürk G, Berillo D, Hedström M, Mattiasson B. Microcontact-BSA imprinted capacitive biosensor for real-time, sensitive and selective detection of BSA. Biotechnology Reports. September 2014, 3: 65–72. PMC 5466099 可免费查阅. PMID 28626651. doi:10.1016/j.btre.2014.06.006. ↩
-
Allender CJ, Richardson C, Woodhouse B, Heard CM, Brain KR. Pharmaceutical applications for molecularly imprinted polymers. International Journal of Pharmaceutics. February 2000, 195 (1–2): 39–43. PMID 10675681. doi:10.1016/s0378-5173(99)00355-5. ↩
-
Ramström O, Skudar K, Haines J, Patel P, Brüggemann O. Food analyses using molecularly imprinted polymers. Journal of Agricultural and Food Chemistry. May 2001, 49 (5): 2105–14. PMID 11368563. doi:10.1021/jf001444h. ↩
-
Sensor Laboratory CNR-IDASC & University of Brescia. Biosensors. Biosensors, SENSOR LABORATORY CNR – IDASC & University of Brescia, Dept. Of Chemistry and Physics. [2012-03-01]. (原始内容存档于2012-04-29). (accessed Feb, 29 2012) ↩
-
Wulff G, Gross T, Schönfeld R. Enzyme Models Based on Molecularly Imprinted Polymers with Strong Esterase Activity. Angewandte Chemie International Edition in English. 1997, 36 (18): 1962. doi:10.1002/anie.199719621. ↩
-
Smolinska-Kempisty K, Guerreiro A, Canfarotta F, Cáceres C, Whitcombe MJ, Piletsky S. A comparison of the performance of molecularly imprinted polymer nanoparticles for small molecule targets and antibodies in the ELISA format. Scientific Reports. November 2016, 6 (1): 37638. Bibcode:2016NatSR…637638S. PMC 5121619 可免费查阅. PMID 27883023. doi:10.1038/srep37638. ↩
-
Olsen J, Martin P, Wilson ID. Molecular imprints as sorbents for solid phase extraction: potential and applications. Anal. Commun. 1998, 35 (10): 13H–14H. doi:10.1039/A806379F. ↩
-
Allender CJ, Richardson C, Woodhouse B, Heard CM, Brain KR. Pharmaceutical applications for molecularly imprinted polymers. International Journal of Pharmaceutics. February 2000, 195 (1–2): 39–43. PMID 10675681. doi:10.1016/s0378-5173(99)00355-5. ↩
-
Bedwell TS, Whitcombe MJ. Analytical applications of MIPs in diagnostic assays: future perspectives. Analytical and Bioanalytical Chemistry. March 2016, 408 (7): 1735–51. PMC 4759221 可免费查阅. PMID 26590560. doi:10.1007/s00216-015-9137-9. ↩
-
Chen J, Shinde S, Koch MH, Eisenacher M, Galozzi S, Lerari T, et al. Low-bias phosphopeptide enrichment from scarce samples using plastic antibodies. Scientific Reports. July 2015, 5: 11438. Bibcode:2015NatSR…511438C. PMC 4486973 可免费查阅. PMID 26126808. doi:10.1038/srep11438. ↩
-
Polyakov MV. Adsorption properties and structure of silica gel. Zhurnal Fizicheskoi Khimii. 1931, 2: S. 799–804. ↩
-
US 3338249,Hans Erlenmeyer,“Filter material for tobacco smoke”,发表于1965-08-29 ↩
-
Turner NW, Jeans CW, Brain KR, Allender CJ, Hlady V, Britt DW. From 3D to 2D: a review of the molecular imprinting of proteins. Biotechnology Progress. 2006, 22 (6): 1474–89. PMC 2666979 可免费查阅. PMID 17137293. doi:10.1021/bp060122g. ↩
-
Lorenzo RA, Carro AM, Alvarez-Lorenzo C, Concheiro A. To remove or not to remove? The challenge of extracting the template to make the cavities available in Molecularly Imprinted Polymers (MIPs). International Journal of Molecular Sciences. 2011, 12 (7): 4327–47. PMC 3155354 可免费查阅. PMID 21845081. doi:10.3390/ijms12074327 可免费查阅. ↩
-
Ellwanger A, Berggren C, Bayoudh S, Crecenzi C, Karlsson L, Owens PK, et al. Evaluation of methods aimed at complete removal of template from molecularly imprinted polymers. The Analyst. June 2001, 126 (6): 784–92. Bibcode:2001Ana…126..784E. PMID 11445938. doi:10.1039/b009693h. ↩
-
Soxhlet, F. “Die gewichtsanalytische Bestimmung des Milchfettes”. Polytechnisches J. (Dingler’s) 1879, 232, 461. ↩
-
Luque de Castro MD, Priego-Capote F. Soxhlet extraction: Past and present panacea. Journal of Chromatography A. April 2010, 1217 (16): 2383–9. PMID 19945707. doi:10.1016/j.chroma.2009.11.027. ↩ ↩2
-
Hillberg AL, Brain KR, Allender CJ. Design and evaluation of thin and flexible theophylline imprinted polymer membrane materials. Journal of Molecular Recognition. 2009, 22 (3): 223–31. PMID 19177493. S2CID 25997199. doi:10.1002/jmr.935. ↩
-
Canfarotta F, Poma A, Guerreiro A, Piletsky S. Solid-phase synthesis of molecularly imprinted nanoparticles. Nature Protocols. March 2016, 11 (3): 443–55. PMID 26866789. S2CID 20963528. doi:10.1038/nprot.2016.030. ↩
-
Poma A, Guerreiro A, Whitcombe MJ, Piletska EV, Turner AP, Piletsky SA. Solid-Phase Synthesis of Molecularly Imprinted Polymer Nanoparticles with a Reusable Template - “Plastic Antibodies”. Advanced Functional Materials. June 2013, 23 (22): 2821–2827. PMC 4746745 可免费查阅. PMID 26869870. doi:10.1002/adfm.201202397. ↩
-
Cintas P, Luche JL. Green chemistry. The sonochemical approach. Green Chem. 1999, 1 (3): 115–125. doi:10.1039/a900593e. ↩
-
Luque-Garcia JL, de Castro L. Ultrasound: A powerful tool for leaching. Trends Anal. Chem. 2003, 22: 90–99. doi:10.1016/S0165-9936(03)00102-X. ↩
-
Ellwanger A, Berggren C, Bayoudh S, Crecenzi C, Karlsson L, Owens PK, et al. Evaluation of methods aimed at complete removal of template from molecularly imprinted polymers. The Analyst. June 2001, 126 (6): 784–92. Bibcode:2001Ana…126..784E. PMID 11445938. doi:10.1039/b009693h. ↩
-
Tobiszewski M, Mechlińska A, Zygmunt B, Namieśnik J. Green analytical chemistry in sample preparation for determination of trace organic pollutants. Trends Anal. Chem. 2009, 28 (8): 943–951. doi:10.1016/j.trac.2009.06.001. ↩
-
Ertürk G, Berillo D, Hedström M, Mattiasson B. Microcontact-BSA imprinted capacitive biosensor for real-time, sensitive and selective detection of BSA. Biotechnology Reports. September 2014, 3: 65–72. PMC 5466099 可免费查阅. PMID 28626651. doi:10.1016/j.btre.2014.06.006. ↩